plotlyの自分用Layoutテンプレート
最近はpandas.DataFrameをプロットするときにplotly.expressを使うことが増えた。
matplotlibよりも系列ごとにsymbol, colorなどを変えるのが手軽なのが気に入っているが、デフォルトのレイアウトだとスライドに貼ったり、人に見せるには微妙だった。
ということで、seaborn.set_context('poster')に寄せたgo.Layout.Templateを作ってみた。
何もしない場合が↓で、
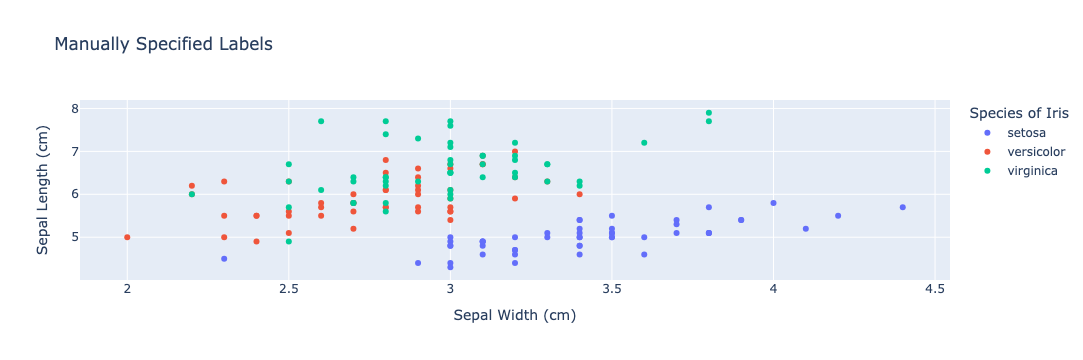
テンプレートを指定した場合は↓の通り。
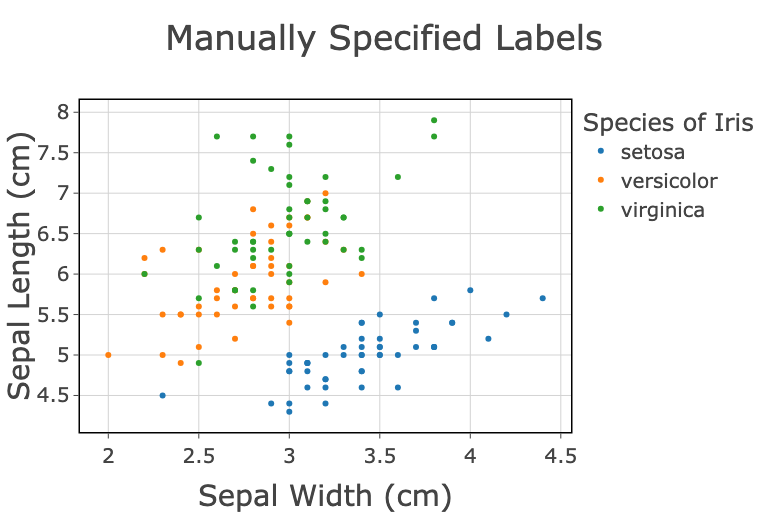
まあこれならpublication readyではないけど、共有用には十分かなといったところ。
References
GitHub pages から他サイトにリダイレクト
やりたいこと
GitHub pages でホストしていたドキュメントをRead the Docs に移行したので、GitHub pages からリダイレクトしたい。 ページ構成は同じなのでサブページもリダイレクトしてほしい:
- https://lan496.github.io/hsnf → https://hsnf.readthedocs.io/en/latest/
- https://lan496.github.io/hsnf/definition.html → https://hsnf.readthedocs.io/en/latest/definition.html
手順
- まず
gh-pagesbranch のファイルを消す
git checkout gh-pages git rm -r *
index.htmlを作って、index page をリダイレクトさせる
<!DOCTYPE html> <head> <meta charset="utf-8"> <title>Redirecting to https://hsnf.readthedocs.io/en/latest/</title> <meta http-equiv="refresh" content="0; URL=https://hsnf.readthedocs.io/en/latest/"> <link rel="canonical" href="https://hsnf.readthedocs.io/en/latest/"> </head> </html>
404.htmlでサブページもリダイレクトさせる
<!DOCTYPE html> <html> <head> <meta charset="utf-8"> <title>404</title> <script> window.onload = function () { if (window.location.href.includes("lan496.github.io/hsnf")) { const redirectLocation = window.location.href.replace( "lan496.github.io/hsnf", "hsnf.readthedocs.io/en/latest" ); document.getElementById("body").innerHTML = "Redirecting to " + redirectLocation + " ..."; window.location.href = redirectLocation; } } </script> </head> <body id="body" style="text-align: center"> Page not found </body> </html>
- push する
git add index.html git add 404.html git commit git push origin gh-pages
これでリダイレクトできる
References
- Redirecting GitHub pages after a repository move https://gist.github.com/domenic/1f286d415559b56d725bee51a62c24a7
- https://gist.github.com/domenic/1f286d415559b56d725bee51a62c24a7?permalink_comment_id=3945701#gistcomment-3945701
- Creating a custom 404 page for your GitHub Pages site - GitHub Docs https://docs.github.com/en/pages/getting-started-with-github-pages/creating-a-custom-404-page-for-your-github-pages-site
NotionでGTDタスク管理2022年版
Notion布教と自分の備忘録を兼ねてBoard viewを使ったタスク管理の現状の運用をまとめておく. 複数人による運用は考えない.半日単位くらいで予定を入れられる人向け.
テンプレートを↓に作成した.以下はNotionの設定のポイントと現状の運用ルール. season-yarrow-b96.notion.site
Database
- できるだけひとつのデータベースにまとめる(研究室用・家用みたいな分割はしない)
Property
“View options→Properties”から以下を設定しておく
- Name (as title, visible): タイトル
- Context (multi-select, visible): “研究”.”家”のようにタグ付けに使う
- Due Date (Date. visible): 締め切り
- URL: 関連URL.GitHub issue のリンクを貼ることが多い
- Status (Select): タスクの状態(Status propertyで後述)
- Last edited time
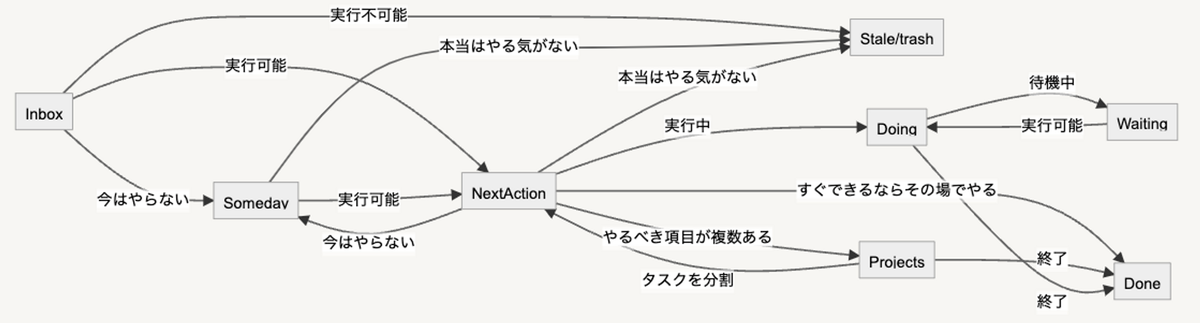
Status property
だいたいGTDに沿ってタスクの状態を分けておく(Ref: https://hamberg.no/gtd)
- Inbox
- 思いついたら兎に角ここにいれる。まだactionableな粒度になっていなくてもよい
- Someday
- 現状actionable にできなさそうなものを入れておく
- Next Action
- タイトルはActionable なものでなければならない
- 時間があるなら今週中に取り組みたいと思えるもの(正直に!)
- Doing
- 実行中のタスク
- Projects
- 複数のタスクをまとめる
- タイトル/overview/expected outcome だけ書いておく
- 対応するタスクが必ず”Doing”に一つ以上ある
- 複数のタスクをまとめる
- Waiting
- 対応待ち・期日になるまで動けない/動く必要がないタスクを置く
- 必ずdue date を設定する
- Done
- 終了したことを保存する
- Stale
- やらなさそうなことを捨てる場所(タスクカードをいきなり削除するのは心理的障壁が高いので)
Board view
- Context ごとにフィルターしたviewをつくるとよい
Timeline view
- FilterでStatusがDoing, Waiting, Next Action, Projects のいずれかのものだけ表示させる
- Due Date で並び替えておく
Workflow
GTDに従って粛々とタスクをこなす.
Weekly review
1週間に一度Board全体を見直して古くなっている部分を更新する.
以下のチェックリストを上から実行していく
- [ ] 机を片付ける
- [ ] カレンダーの予定を確認
- [ ] 今週の日記を見返す: まとまった情報をメモに移動
- [ ] Inbox を空にする
- [ ] Review Action Lists: “Next Action”にいるけど実際にはやる気のないものをSome DayまたはStaleに移動
- [ ] Review Project Lists: Projectに対応するactionがDoingにいる
- [ ] Review Some-Day Lists: Next Actionに移せるものがあるか
- [ ] Done に入ったタスクに書いた内容をメモに移動
Notion tips
@nowで現在時刻を入力できる@todayで今日の日付を入力できる- toggle heading
- Notion Boost https://chrome.google.com/webstore/detail/notion-boost/eciepnnimnjaojlkcpdpcgbfkpcagahd/related で以下を有効化
- Show Outline
- Full width for all pages
- ‘Scroll to top’ button
- Close slash command menu after space
- Left align media
- Hide Help button from pages
- 定期的なタスクはZapierと連携して作成できる(が,Trelloに比べると微妙)
- https://blog.deepblue-ts.co.jp/web/zapier_notion_slack_reminder/
- 現状ではWeekly review とバックアップのタスクを定期的に作らせている
Spglibに磁気空間群関連の機能を追加した
先日リリースされたspglib 2.0 から自分の実装した磁気空間群の判定・結晶構造のstandardizationが追加された。
- リリースノート: Summary of releases — Spglib v.2.0.0
- C API: C-APIs — Spglib v.2.0.0
- Python API: Spglib for Python — Spglib v.2.0.0
miniMD/LAMMPSのMPI周りを読んだ
miniMDというLAMMPSの開発チームが作っている軽量(.hと.cpp合わせて6500行程度)なMDコードを眺めた。
まずminiMD/LAMMPSのMPI並列のデザインをまとめてから、miniMD中の該当するコードを参照していく。
Parallel algorithm in miniMD/LAMMPS
Spatial decomposition
https://docs.lammps.org/latest/Developer_parallel.html#parallel-algorithms
LAMMPSではspatial decomposition に基づいてMPI分散する。simulation box を空間的に分割して、それぞれのsub-domainを各プロセスが担当する。
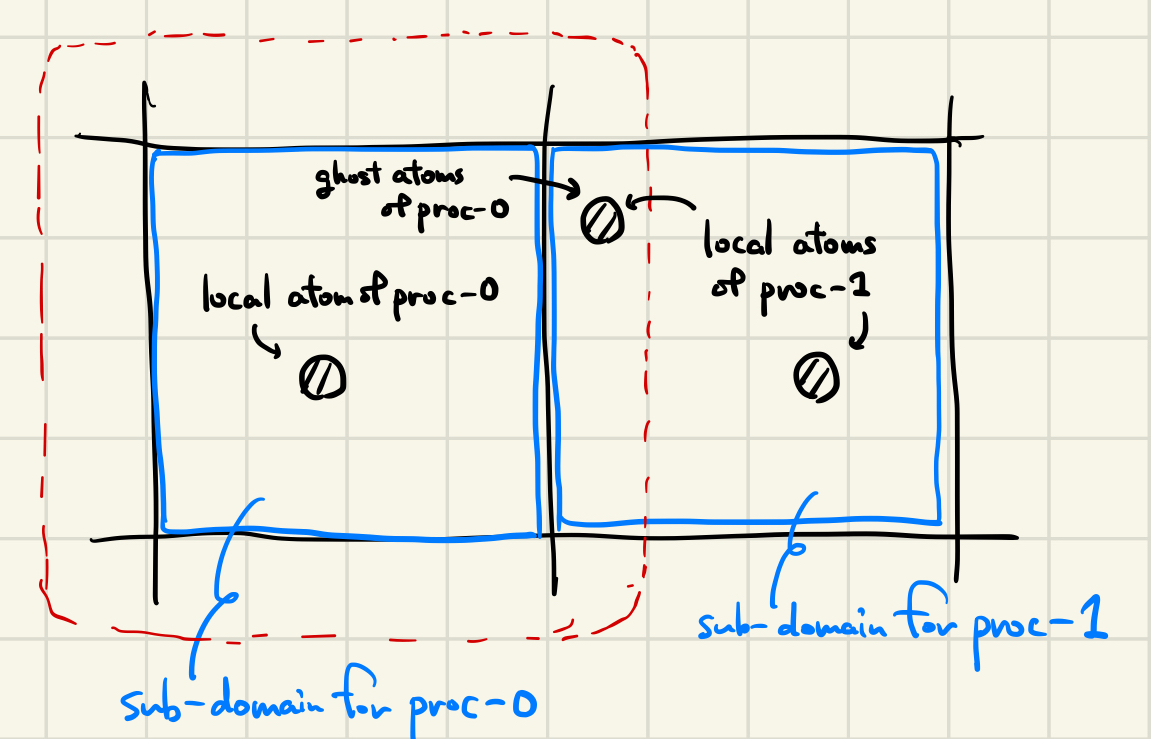
そのプロセスの担当するsub-domain にいる原子をlocal atom と呼ぶ(LAMMPSのドキュメントではowned atom と呼んでいるがコード内ではlocal atom と呼ばれている)。 ただし、各プロセスはlocal atom だけを保持しているのではなく、sub-domain の端からポテンシャルのカットオフ半径(から少し伸ばした距離)だけ広げた範囲に含まれる原子の情報も保持している。 この追加分の範囲に含まれる原子(隣のsub-domain を担当するプロセスにとってのlocal atom)はghost atom と呼ばれる。 上図では赤枠部分がプロセス0の追加分の範囲で、真ん中の原子はプロセス1にとってlocal atom として、プロセス0にとってはghost atom として、両方のプロセスが情報を保持する。
Communication pattern
https://docs.lammps.org/latest/Developer_comm_ops.html#communication-patterns
タイムステップごとに原子位置は変化するのでプロセス間で原子位置をMPI通信する必要がある。 これはforward communication と呼ばれている。
また、ghost atom に対して計算した情報をその原子を担当しているプロセスに送りたいことがある。 これはreverse communication と呼ばれている。 例えば、ghost atom jからlocal atom iへのpair force fijを計算したときに、fji は符号を反転するだけで得られるのでghost atom j にfjiを覚えさせて通信すれば演算回数を減らせる。
その他、neighbor list を更新するときにする処理はexchange とかborderと呼ばれている。
miniMD code reading
Integration
https://docs.lammps.org/latest/Developer_flow.html#how-a-timestep-works
miniMD/integrate.cpp at ab40f4c8b0808959abe1e60508e698d6d0d65ac1 · Mantevo/miniMD · GitHub
integrate.cppに各ステップごとにやることが書いている(LAMMPSのverlet.cppに対応)。とりあえずここから読み始めるとよい。
以下は擬コード
for(n = 0; n < ntimes; n++) { initialIntegrate(); if((n + 1) % neighbor.every) { comm.communicate(atom); // Forward communication of atom positions } else { // Rebuild neighbor lists comm.exchange(atom); // Send atoms outside box to a proper process comm.borders(atom); neighbor.build(atom); } force->compute(atom, neighbor, comm, comm.me); // Calculate forces if(neighbor.halfneigh && neighbor.ghost_newton) { comm.reverse_communicate(atom); // Send pair forces between local and ghost } finalIntegrate(); if(thermo.nstat) { thermo.compute(n + 1, atom, neighbor, force, timer, comm); } }
Communication in miniMD
Forward communication
送るlocal atom の原子位置をdouble配列に詰めてMPI_Sendrecvで送り合っている。
miniMD/atom.cpp at ab40f4c8b0808959abe1e60508e698d6d0d65ac1 · Mantevo/miniMD · GitHub
miniMD/comm.cpp at ab40f4c8b0808959abe1e60508e698d6d0d65ac1 · Mantevo/miniMD · GitHub
Reverse communication
同様に送るghost atom のforce をdouble配列に詰めてMPI_Sendrecvで送り合っている。
miniMD/atom.cpp at ab40f4c8b0808959abe1e60508e698d6d0d65ac1 · Mantevo/miniMD · GitHub
miniMD/comm.cpp at ab40f4c8b0808959abe1e60508e698d6d0d65ac1 · Mantevo/miniMD · GitHub
Neighbor list and newton pair
https://docs.lammps.org/latest/Developer_par_neigh.html#neighbor-lists
neighbor list に含まれる原子はneighbor flag とnewton flag によって決まる。
Half neighbor list (neighbor=half) with newton=off
miniMD/force_lj.cpp at ab40f4c8b0808959abe1e60508e698d6d0d65ac1 · Mantevo/miniMD · GitHub
あるプロセスのlocal atom i からみてカットオフ半径内に原子jがいるとき
- j がlocal atom ならば、(i, j)の組は原子iのneighbor list と原子jのneighbor list のどちらか一方にしか出てこない
- j がghost atom ならば、(i, j)の組は原子iのneighbor list と原子jのneighbor list の両方に含まれる(原子j のneighbor list は原子jがlocal atom であるプロセスが保持している)
以下はpotential energyとforce を計算する擬コード。 ポテンシャルを実装するときは、やってくるneighbor list に合わせて実装を書く必要がある。
for i in local atoms of process-p: fi = 0 for j in half neighbor list of atom-i: Compute fij (forces from atom-j to atom-i) fi += fij if j is local atom of process-p: f[j] -= fij Compute eij if j is local atom of process-p: eng_vdwl += eij else: eng_vdwl += eij / 2
Half neighbor list (neighbor=half) with newton=on
miniMD/force_lj.cpp at ab40f4c8b0808959abe1e60508e698d6d0d65ac1 · Mantevo/miniMD · GitHub
あるプロセスのlocal atom i からみてカットオフ半径内に原子jがいるとき
ghost atom jに足されたforce はreverse communication で対応するプロセスに送られる。 逆に、newton=onにしないとreverse communication が呼ばれない。 LAMMPSでも同様。
for i in local atoms of process-p: fi = 0 for j in half neighbor list of atom-i: Compute fij (forces from atom-j to atom-i) fi += fij f[j] -= fij Compute eij eng_vdwl += eij
Full neighbor list (neighbor=full)
miniMD/force_lj.cpp at ab40f4c8b0808959abe1e60508e698d6d0d65ac1 · Mantevo/miniMD · GitHub
あるプロセスのlocal atom i からみてカットオフ半径内に原子jがいるとき
意味的にはneighbor=fullならnewton=off(反作用の計算はしない)だが、ポテンシャルの計算をする部分でnewton flag は必要ない。
ただし、機械学習ポテンシャルを含むmany-body potential の場合、原子iのforceを計算するためにはカットオフ半径中の原子jのfull neighbor list が必要になることがある。 この場合、reverse communication で原子iのforceの一部を送る必要があるので、newton=onに設定しなければいけない1。
for i in local atoms of process-p: fi = 0 for j in full neighbor list of atom-i: Compute fij (forces from atom-j to atom-i) fi += fij Compute eij eng_vdwl += eij f[i] += fi
-
”half neighbor list の中身を決めるフラグ”と”reverse communicationを実行するかのフラグ”が使いまわされている。↩
Materials Project new API 関連のリンク
去年の11月からMaterials Project API が新しくなったみたい。
今までのfrom pymatgen.ext.matproj import MPResterでpython client を呼ぶのはdeprecate されて、代わりにmp-apiをpip install して
from mp_api import MPRester
せよ、とのこと。
https://next-gen.materialsproject.org/api docs.materialsproject.org
REST API 一覧のページはあるけれど、python client との対応がよくわからないので、ソースコードを読むのが一番早いと思う。