点群・空間群の既約表現ライブラリを作った+私的Python開発環境2022
既約表現は結晶の対称性を扱うときに強力なツールだが、自分の用途に使えそうなライブラリを見つけられなかった1。 ということで作った:
実装したアルゴリズムや使い方はドキュメントに書いたので、ここでは実装(python)の環境を忘れないうちに書き残しておく。
Pythonパッケージ開発環境
以下、最近使っているPythonのパッケージング周りのライブラリのリンクと雑感
setuptools
pyproject.toml だけで設定できるらしいと聞きつつもsetup.pyを併用している。
Optional dependency (setup.pyならextra_require)を設定しておくと
pip install -e .[dev,doc]
みたいな感じで必須ではない依存ライブラリを管理できてよい。
setuptools-scm
git tag からversion stringを生成してくれる。
pre-commit
black, flake8, mypy, isort など有名なやつは最低限入れている。
今回はjupyter notebook にリンターをかけるためにnbQAを使ってみたがかなり便利だった github.com
pre-commit.ci も設定しておいて損はない。 pre-commit.ci
テスト
pytest一択
ドキュメント
定式化がバックグラウンドにあるときは、まず実装メモを書いてそれを読みながら実装するのがベストプラクティスだと思っている。 このときメモと実装が近ければ近いほどstaleしづらいので、自分は実装メモも実装と同じレポジトリで管理している。
ドキュメント自体はsphinxで生成しているが、rstはつらいのでmarkdown っぽく書ける拡張を使っている。 myst-parser.readthedocs.io
素のmarkdownだと論文の引用を書くのが面倒だが、sphinxの拡張を入れるとbibtexを読めるようになる。
Sphinxのテーマはsphinx-book-themeを使っている。前はfuroを使っていた。ここらへんは好みで。
Github actions
spgrep/deploy.yml at 14ba9c01931e5da24ff5943d5c162fc3fb1acc45 · spglib/spgrep · GitHub
Sphinxで生成したドキュメントをgh-pagesにデプロイするのはactions-gh-pagesで自動化している。
PyPIにアップロードするのもgh-action-pypi-publishで自動化している。
これらのリリース時のworkflowはgit push origin --tag したときだけ走るように設定している。
-
電子状態の既約表現を求める前提で書かれていたり、MITなどのライセンスと組み合わせられないものだったり。↩
LAMMPSで格子変形を許して構造緩和をする
LAMMPSで格子変形を許して構造緩和をさせる(fix 1 box/relax tri 0を指定してminimize commandを実行する)とき、初期構造が著しく歪んでいるせいで一度minimizeさせるだけでは収束しきらないことがある。
このケースにハマっている人をたまに見るので、収束させるためのスクリプトの書き方を以下のnotebookにまとめた。
ポイントとしては
- 一度に全ての自由度を動かさない(例えば、原子位置->原子位置+体積->原子位置+格子の順に
minimizeしていく) ftol(force vectorのL2ノルム)が期待通り小さくなっていることを確認するmaxiterの上限いっぱいまでiterationが回っていないことを確認する
くらいに気をつけるといいと思う。
nglviewのpymatgen wrapperをつくった
以前、nglviewで結晶構造をjupyter notebook上で表示させた続き。
nglviewで原子位置を表示させることは簡単にできるが、ボンドや配位多面体が表示されないなど微妙な点があった。 ということで自分用にnglviewの機能を使ってpymatgenのStructureをいい感じに描画するための小さいライブラリを書いた。
JmolとCrystal Toolkitの表示が個人的に好みなのでそれらに寄せた表示をするようにしている。
Nsight Systems and Nsight Compute
Nsight Tools composes of Nsight Systems and Nsight Compute 1. Nsight Systems profiles a whole application (for Pascal and newer). Nsight Compute profiles a CUDA kernel (for Volta and newer).
Environment
- Host
- Ubuntu 20.04LTS
- Remote on Singularity
- Singularity==3.8.1
- Debian-based container:
From: nvidia/cuda:11.2.1-devel-ubuntu20.04
Install Nsight Tools on host
- Nsight Systems: https://developer.nvidia.com/nsight-systems
- Nsight Compute: https://developer.nvidia.com/nsight-compute
After installation, set up PATH:
export PATH=${PATH}:/path/to/NVIDIA-Nsight-Compute
export PATH=${PATH}:/path/to/nsight-systems-2021.4.1/bin
Install Nsight Tools on singularity container (remote)
Set up proper perf_event_paranoid on the remote machine:
sudo sh -c 'echo 2 >/proc/sys/kernel/perf_event_paranoid' sudo sh -c 'echo kernel.perf_event_paranoid=2 > /etc/sysctl.d/local.conf'
Append the following in a Singularity definition file:
# container.sif
%post
# Nsight Systems 2021.4.1 and Nsight Compute 2021.3.0
apt-get update -y
DEBIAN_FRONTEND=noninteractive apt-get install -y --no-install-recommends \
apt-transport-https \
ca-certificates \
gnupg \
wget
rm -rf /var/lib/apt/lists/*
wget -qO - https://developer.download.nvidia.com/devtools/repos/ubuntu2004/amd64/nvidia.pub | apt-key add -
echo "deb https://developer.download.nvidia.com/devtools/repos/ubuntu2004/amd64/ /" >> /etc/apt/sources.list.d/nsight.list
apt-get update -y
DEBIAN_FRONTEND=noninteractive apt-get install -y --no-install-recommends \
nsight-systems-2021.4.1 \
nsight-compute-2021.3.0
rm -rf /var/lib/apt/lists/*
%environment
export PATH=${PATH}:/opt/nvidia/nsight-compute/2021.3.0/
Grant SYS_ADMIN privilege to user on the remote machine:
sudo singularity capability add --user user CAP_SYS_ADMIN
Profile with Nsight Systems on remote
Run container with SYS_ADNIN privilege
singularity run --nv --add-caps=SYS_ADMIN container.sif
Profile a.out and save its report at out.qdrep :
nsys profile --force-overwrite=true --stats=true -o out.qdrep ./a.out
Display profiling report by Nsight Systems on host
Launch Nsight Systems' GUI
nsys-ui
Open out.qdrep, and then you will see a display like below:

On GUI, right-click a kernel and click "Analyze the Selected Kernel with Nsight Compute". Then, a command for profiling the selected kernel with Nsight Compute is displayed like below:
# for example ncu --kernel-name cuda_parallel_launch_constant_memory --launch-skip 10 --launch-count 1 "./a.out"
Profile a kernel with Nsight Compute on remote
Profile and save its report at report.ncu-rep (append -o report -set full -f from the above command):
ncu --kernel-name cuda_parallel_launch_constant_memory --launch-skip 10 --launch-count 1 -o report --set full -f "./a.out"
Display profiling report by Nsight Compute on host
Launch Nsight Compute's GUI
ncu-ui
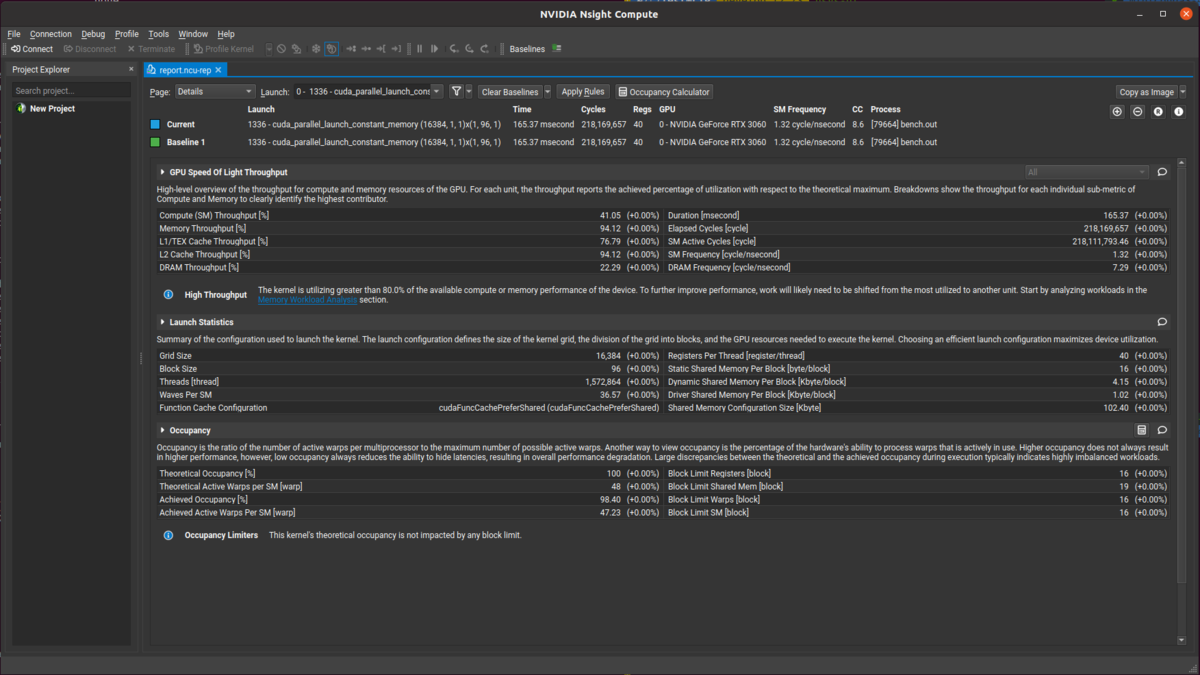
Open report.ncu-rep, and then you will see a display like below:
Misc
Use nvprof
nsys nvprof ./a.out
References
- Nsight Systems
- Nsight Compute
-
“Nsight” seems to be pronounced N-sight↩
matplotlibでfontが見つからないとき
matplotlibでrcParams['font.family'] = 'serif'と設定しているのに
font family ['serif'] not found. Falling back to DejaVu Sans
とエラーが出てserif体が使えないときは、以下のようにキャッシュを消してmsttcorefontsを入れると直る。
rm -r ~/.cache/matplotlib sudo apt install msttcorefonts -qq
https://github.com/matplotlib/matplotlib/issues/13139/ https://stackoverflow.com/questions/42097053/matplotlib-cannot-find-basic-fonts/49884009#49884009
matplotlib≥3.4からはこんなことをしなくてもよくなるらしい。 https://github.com/matplotlib/matplotlib/pull/17521
JAX/HaikuでCrystal Graph Convolutional Neural Network (CGCNN)を実装した
Crystal Graph Convolutional Neural Network (CGCNN)
CGCNNは結晶にGraph Neural Network (GNN)を適用した(おそらく)初めての手法。GNNがやたら流行っている有機分子と違って、結晶の場合はそもそもグラフの作り方が自明でない、結晶の周期性のせいで隣接行列を陽に持てないなどの問題があるが、この手法はそのあたりに取り組んだ点で新しかったのだと思う。手法の詳細については論文を読んでもらうとして、今回はCGCNNのpytorch版公式実装をjax/haikuで書き直したので、ハマった点とメモを残しておく。
JAX
JAXは雑に言うと自動微分とJITコンパイラが使えてバックエンドにGPUを指定できるNumpyみたいなやつ。去年くらいから流行っている気がする。最近はMDとかDFTをJAXで実装する話があって面白いなと思っている1。
- https://roberttlange.github.io/posts/2020/03/blog-post-10/
- https://gcucurull.github.io/deep-learning/2020/04/20/jax-graph-neural-networks/
- https://gcucurull.github.io/deep-learning/2020/06/03/jax-sparse-matrix-multiplication/
- https://sjmielke.com/jax-purify.htm
実装
CGCNNを実装したレポジトリには以下のリンクから飛べる。学習モデルの部分にはdm-haikuを使った。
データセットの収集
公式実装がデータセットに使ったMaterials Project上のmp-idを公開してくれていたので、そこからAPIを叩いてデータセットを集めた。ただ一部の物質でmaterial_idが変更されたらしくて大分ハマった。
前処理
結晶からグラフをつくるのには(元実装と同じく)pymatgen.core.Structure.get_all_neighborsを使った。最初はpymatgen.analysis.graphs.StructureGraphを使ってグラフの作り方をいろいろ試せるように書こうとしていたが、やたら重いので止めた。
ちなみにStructureGraphが重いのは自分が過去に投げたPRのせいで、pymatgen.analysis.local_env.NearNeighborsで隣接している各サイトの属するユニットセルの番号(jimage)を計算する部分がO(num_sites2)になっているから。なんでこんな実装になっているかというと、元の結晶のfractional coordinatesが[0, 1)に収まっていないエッジケースに対応する方法がこれしか思いつかなかったからだ(この修正をしたときにまさかGNNの前処理にこの部分が使われ得るとは思ってもいなかった)。上手い実装が思いつく人はPRを投げてください。
Pooling layer
モデルの実装はpytorch版とほぼ同じように書けるが、graph aggregation の部分だけは注意が必要。ひとつのバッチには次数の異なるグラフたちが並んでいるのでpoolingするのは次数に応じてsplitする必要がある。しかしjax.numpy.split(ary, indices_or_sections, axis)のindices_or_sectionsには(jitするなら)定数しか渡せないし、無理やりstatic_argnumsに突っ込んでもバッチごとにjit recompileされて激重になる。解決策としてはsegment_sumを使えばjitできるようになる。
hyperparameter
argparseでhyperparameterを弄るのが好きでないのでconfig用のdataclassを作ってdataclass-jsonでJSONをパースすることにした。
ベンチマーク
元のpytorch実装と同じhyperparameterで論文と同じデータセットでformation energyを学習させたらMAEが116 meV/atomのモデルを作ることをできた。しかし、論文では39 meV/atomまで下がると言っているので完全には結果を再現できていない。自前で用意したデータセットで元実装を学習させたら90 meV/atomで、これでも論文の値より大きいのでhyperparameter tuningとかの問題なのか?
Future works
いろいろやれることが思いつくけど、モチベが続かない気がするのでとりあえず区切りとする。
- jax-mdとの連携: 夢がある。現状pymatgenでグラフを作る部分が無視できない重さなので粒子の隣接リストもjaxで書く必要がありそう。
- 頂点の次数を可変にする: 元実装と自分の実装ではグラフの各頂点の次数が同じになるようにpaddingしている。これに関してはこことかここで議論されている。
-
既存のコードを置き換えるとは思えないが↩
Materials Projectのmaterial_idとtask_id
Materials Project に登録されているデータはmaterial_id(mp-****みたいなやつ)で指定される。だが、2018年の半ばからmaterial_idが一部の結晶では変更されたようなので注意が必要。
それぞれの計算系にはmaterial_idが振られている。そして各計算系には(single-shotとか構造緩和とかの)複数の計算条件(task_id)が対応している。material_idはtask_idの中で一番精度の高い構造緩和に対応するものが選ばれる(らしい)。で、2018年にデータベースがアップデートされて、それに伴って一部のmaterial_idが以前とは違うtask_idを指すようになった。
例えばCs2CrH2Cl5O(material_id=mp-542535)は以前はmp-633688がmaterial_idとして使われていた。Materials ExplorerでみるときはどちらのIDを使っても同じページにリダイレクトされるので問題ないが、古い方のIDしか知らないときにAPIを叩きたい時は以下のようにする必要がある。
m = MPRester() material_id_old = 'mp-633688' material_id_new = m.get_materials_id_from_task_id(material_id_old) # -> 'mp-542535' # get property formation_energy_per_atom = m.get_data(material_id_new, prop='energy_per_atom') # get structure structure = m.get_structure_by_material_id(material_id_new)
参考
- https://matsci.org/t/significant-release-of-new-database-schema-and-additional-data/1259/7
- https://matsci.org/t/change-in-materials-project-ids/1268/2
- https://matsci.org/t/merge-of-material-id/1631/2
- https://matsci.org/t/some-entries-seem-not-to-be-queryable/1279/2
- https://pymatgen.org/pymatgen.ext.matproj.html